CVPR (Computer Vision and Pattern Recognition) 是计算机视觉和模式识别领域的国际顶级学术会议,在中国计算机学会推荐国际期刊和会议中,CVPR为人工智能领域的A类会议。近期,人工智能学院教授张道强课题组4篇论文入选CVPR 2025。
论文1:视觉-语言模型的知识产权保护方法研究
2023级博士生汪恋雨,在新加坡科技研究局研究员付华柱以及张道强的指导下以第一作者发表了论文《Vision-Language Model IP Protection via Prompt-based Learning》。论文提出了IP-CLIP,一种专为CLIP设计的轻量级知识产权保护策略。该方法通过提示学习和CLIP的冻结视觉骨干提取图像风格和内容信息,形成防止特征非法迁移的屏障。此外,引入风格增强分支,构建授权和非授权域的特征库,进一步加强保护能力。研究团队还提出了三项新指标,旨在更好地平衡授权域与非授权域之间的性能衰减。实验结果表明,IP-CLIP在多个场景下表现出色,为VLMs的知识产权保护提供了创新解决方案。
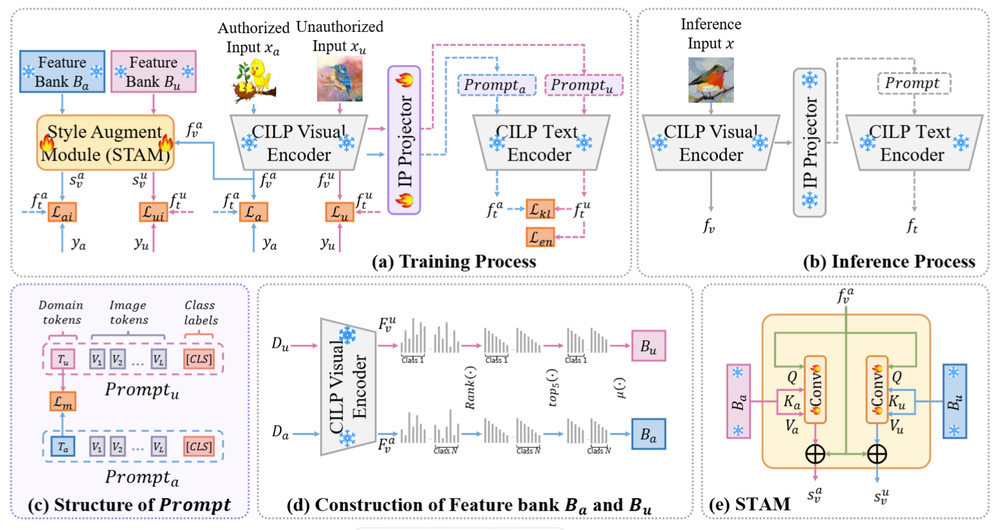
图1 IP-CLIP框架展示图

图2不同模型性能可视化比较
论文2:基于多模态拓扑嵌入图学习的基因预测方法
2023级硕士生石航,在人工智能学院副教授邵伟的指导下,以第一作者发表了论文《Multi-modal Topology-embedded Graph Learning for Spatially Resolved Genes Prediction from Pathology Images with Prior Gene Similarity Information》。该论文提出一种基于多模态拓扑嵌入图学习的基因预测方法。该方法由三个核心模块构成:多模态图嵌入模块、空间邻域排序模块及基于先验基因本体知识的图神经网络模块。多模态图嵌入模块通过深入学习不同模态内部及模态间的特征交互,生成全面的图像块表示,从而增强特征的表达能力和判别性。空间邻域排序模块则通过保持节点间的空间拓扑关系,确保与目标节点空间距离较近的邻域节点在特征空间中的相似度得到加强,进而有效捕捉局部和全局空间依赖。基于先验基因本体知识的图神经网络模块通过挖掘基因间的功能相似性,结合基因本体的先验信息,优化基因表达的预测精度,从而提高模型的生物学推理能力。实验结果表明,M2TGLGO在多个公开数据集上表现出显著优于现有方法的预测精度,展示了其在空间转录组学数据分析中的广泛应用潜力。
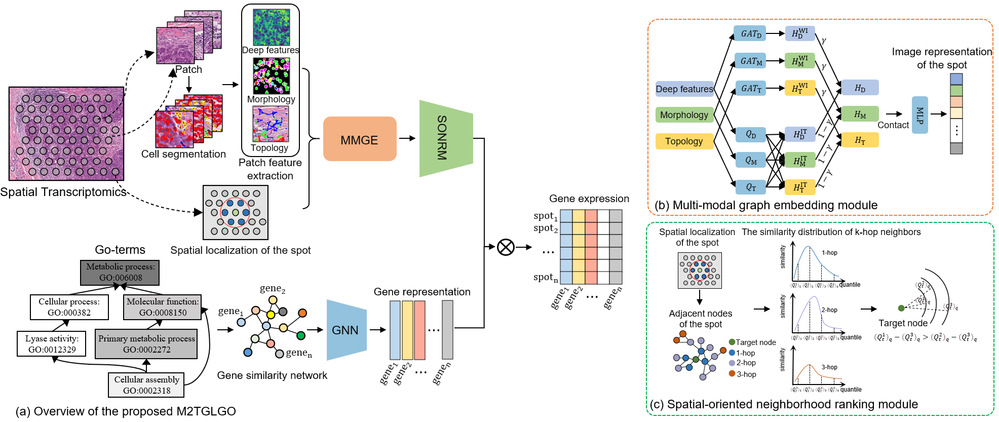
图3 M2TGLGO框架展示图

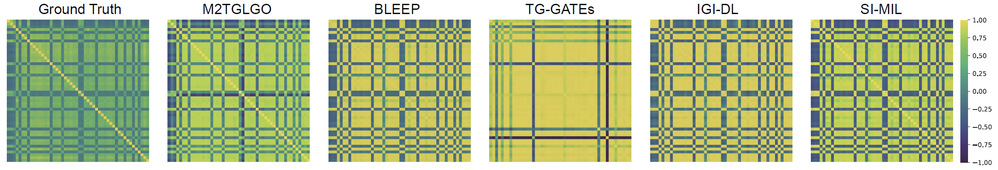
图4 不同模型性能比较
论文3:基于潜在分化条件变分自编码器的鲁棒多模态生存预测
2024级博士生周俊杰,在邵伟和张道强的指导下,以第一作者发表了论文《Robust Multimodal Survival Prediction with the Latent Differentiation Conditional Variational AutoEncoder》。该论文提出了一种基于潜在分化条件变分自编码器(LD-CVAE)的鲁棒多模态生存预测方法,旨在解决基因组数据缺失情况下的癌症生存预测问题。针对现有方法通常假设所有模态数据完整可用,而实际中基因组数据采集成本高、测试样本中可能缺失的问题,该方法通过从病理图像生成基因组表示来应对数据不完整性。然而,这一策略面临两大挑战:(1)全病理切片图像(WSIs)数据量大,难以有效表示;(2)在统一的生成框架下生成具有多样化功能类别的基因组嵌入具有挑战性。研究提出通过变分信息瓶颈Transformer模块从WSIs中学习压缩的病理表示,并利用潜在分化变分自编码器生成具有不同功能的基因组特征。最后,采用product-of-experts将基因组后验和图像后验整合,用于联合潜在分布估计。在五个不同的癌症数据集上的实验结果表明该方法在完整模态和缺失模态场景下均表现出优越性,验证了其有效性和鲁棒性。

图5. LD-CVAE算法流程图
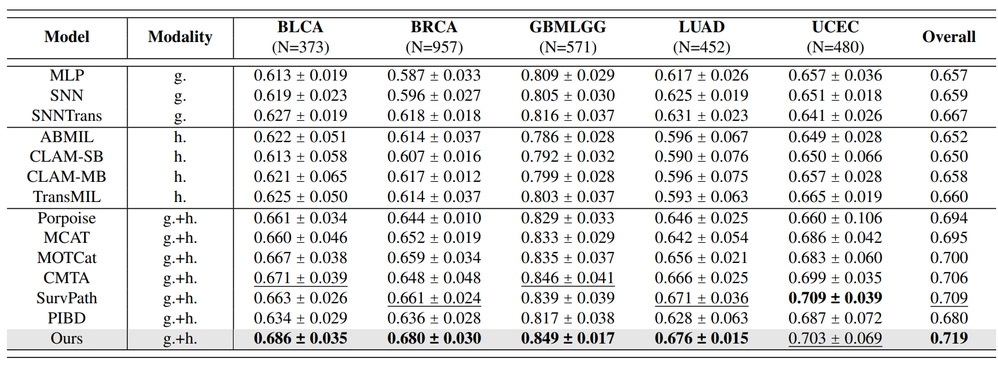
表1 不同方法一致性指标比较

图6. 不同方法生存分析曲线比较
论文4:基于发散感知多模态扩散模型的纳米颗粒分布预测
2024级博士生周俊杰,在南京医科大学教授王守巨、邵伟和张道强的指导下以第一作者发表了论文《DAMM-Diffusion: Learning Divergence-Aware Multi-Modal Diffusion Model for Nanoparticles Distribution Prediction》。该论文提出了一种发散感知多模态扩散模型,用于自适应地生成单模态和多模态分支的预测结果,以解决纳米颗粒(NPs)分布预测中的关键问题。纳米颗粒分布在肿瘤诊断和治疗中具有重要意义,而肿瘤微环境的异质性高度影响了NPs在肿瘤中的分布。然而,多模态TME组件之间的分布差异可能导致副作用,例如单模态模型可能优于联合生成模型。为此,研究团队提出了DAMM-Diffusion模型,通过统一网络结合单模态和多模态分支进行预测。具体而言,单模态分支采用U-Net架构,而多模态分支通过引入多模态融合模块MMFM和不确定性感知融合模块UAFM进行扩展。MMFM用于融合多模态特征,UAFM则通过学习不确定性图进行跨注意力计算。随后,发散感知多模态预测器模块评估多模态数据与不确定性图的一致性,以决定最终预测结果来自多模态还是单模态分支。实验结果表明,在给定肿瘤血管和细胞核的TME组件情况下,DAMM-Diffusion能够以更高的准确性生成NPs分布,优于对比方法。
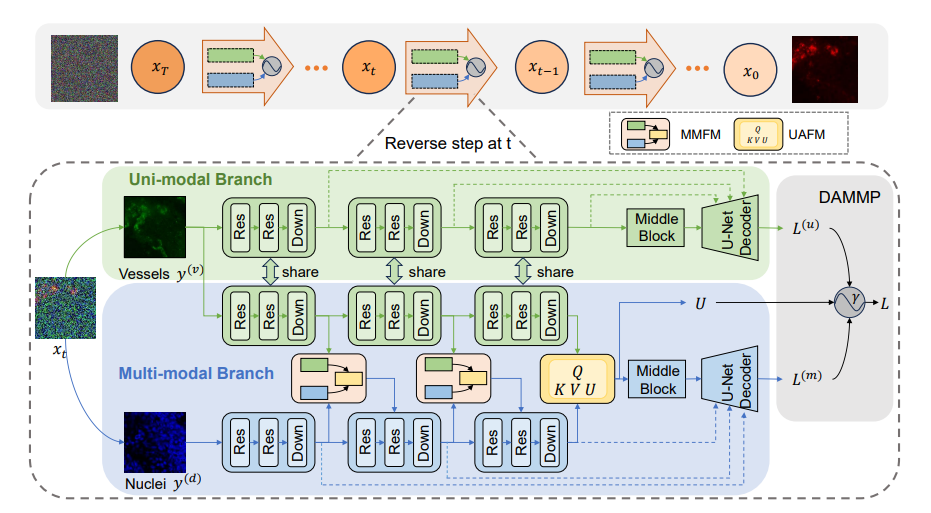
图7 DAMM-Diffusion 算法流程图
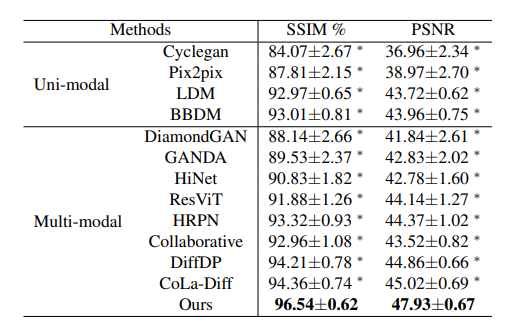
表2 不同方法在SSIM, PSNR指标上的比较
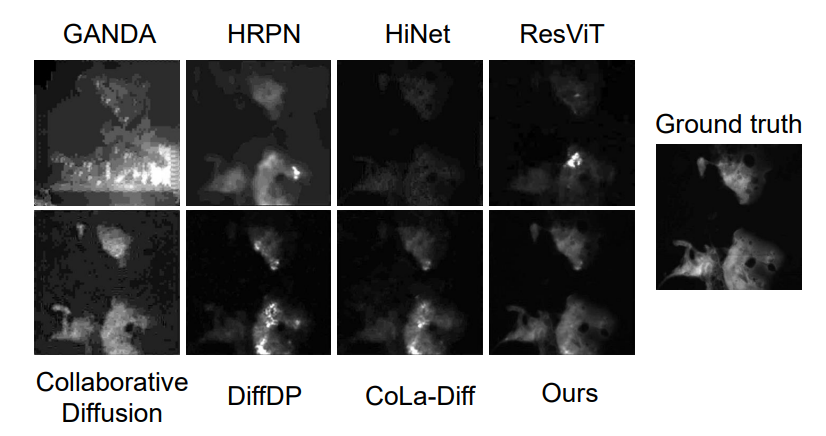
图8 不同方法可视化结果比较